染色体免疫共沉淀与下一代测序结合(ChIP-seq)可以分析细胞或组织的全基因组表观遗传谱,但ChIP-seq这个技术需要大量的细胞来获得高质量的数据,与RNA-seq, Genome DNA-seq技术相比获得核酸的步骤更加复杂,需要进行免疫沉淀实验获得核酸,这局限了ChIP-seq应用于稀有细胞的研究。目前已有的低细胞量的ChIP-seq方法只是在获得核酸后进行扩增以满足第二代测序文库构建的需求,这无法避免ChIP实验中大量核小体信息的丢失,同时引起数据真实度下降,不利于后续的分析。
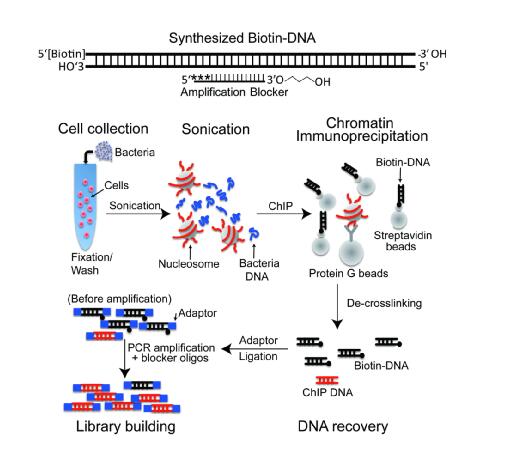
贾俊岭和郑诣先课题组共同开发了RP(Recover via Protection)-ChIP-seq技术以解决ChIP与测序文库构建中信息丢失的问题,从而获得高质量的表观遗传谱。使用与哺乳动物相差较远的酿酒酵母核小体保护ChIP实验中细胞的核小体,并一起分离,获得的核酸量可满足二代测序建库的要求,避免了扩增引起的偏倚出现。针对酵母缺乏许多表观遗传修饰,酵母DNA在文库中会增加测序成本这两个问题,两个课题组又携手开发了FARP(Favored Amplification Recover via Protection)-ChIP-seq技术,可满足极低细胞量细胞的所有组蛋白修饰与转录因子ChIP-seq实验,与此同时可将测序的成本控制在最低。
利用RP-ChIP-seq技术,研究人员展示了鼠晶状体衰老过程中全基因组表观遗传谱的改变,发现表观遗传谱中相异的位点与已知的晶状体衰老相关基因具有一致性;进而,利用FARP-ChIP-seq技术分析了鼠造血干细胞系中的LT-HSC,ST-HSC与MPP细胞的异同,揭示出造血干细胞系丢失了二价基因的存在以满足造血干细胞谱系发生。
这套技术可高保真的展示出任何微量细胞的全基因组表观遗传谱量,为揭示发育过程的表观遗传学机制奠定了基础。
浙江大学生命科学研究院贾俊岭教授与卡内基胚胎研究所的郑诣先研究员为本文的共同通讯作者。




